Das Umweltlabor der Durchflusszytometrie-Gruppe ist auf die umfassende Charakterisierung einzelner Zellen in mikrobiellen Populationen oder komplexen mikrobiellen Gemeinschaften spezialisiert. Hierbei werden sowohl die zelluläre Heterogenität reiner Populationen als auch dynamische Veränderungen in der Zusammensetzung von Gemeinschaften über die Zeit routinemäßig analysiert. Die gewonnenen Daten sind entscheidend für das Verständnis der Prinzipien der Populations- und Gemeinschaftsökologie und ermöglichen die Bestimmung von Wachstumsraten indigener Zelltypen. Parallel zu den Lichtstreuungseigenschaften können autofluoreszierende Pigmente und Proteine pro Zelle quantifiziert werden. Darüber hinaus können Vitalitätszustände oder andere Zelleigenschaften, die extrinsisch mit fluoreszierenden Farbstoffen angefärbt wurden, zur Bestimmung verschiedener physiologischer Zustände herangezogen werden. Zellen aus nahezu jeder Umgebung können in unserem Umweltlabor analysiert werden. Für die Auswertung dieser Daten stehen fortschrittliche bioinformatische Tools zur Verfügung.
Ein weiterer entscheidender Vorteil ist die Möglichkeit der fluoreszenzaktivierten Zellsortierung (FACS). Diese Technologie ermöglicht es, Zelluntergruppen mit unterschiedlichen physiologischen Zuständen oder spezifische Zelltypen aus Gemeinschaften zu separieren. Dies ebnet den Weg für weiterführende Analysen wie Sequenzierung (16S rRNA-Gen-Sequenzierung oder Gesamtgenom-Sequenzierung) oder proteomische Untersuchungen. Zellen können auch in definierten Mengen (zwischen 1 und 1000) in Well-Plates zur nachfolgenden Kultivierung oder auf mikroskopischen Objektträgern zur Visualisierung gezählt und sortiert werden.
Prinzip der Durchflusszytometrie in der Umweltanalytik
Die Durchflusszytometrie ist eine auf UV- und sichtbarem Licht basierende mikroskopische Technologie, die eine multiparametrische Analyse von Zellen ermöglicht, während diese einzeln in einem Flüssigkeitsstrom an einem Objektiv vorbeifließen. Die hydrodynamische Fokussierung gewährleistet dabei, dass die Zellen präzise ausgerichtet und als Einzelzellen für eine genaue Analyse bereitgestellt werden. Während die Zellen den Detektionsbereich passieren, werden sie von Photonen getroffen, die von einem oder mehreren Lasern emittiert werden. Die Zellen emittieren oder streuen Photonen, die dann von Photomultiplier-Röhren (PMTs) erfasst werden. Diese erzeugen einen verstärkten elektrischen Strom, der mit den Fluoreszenzintensitäten oder Streuungseigenschaften der Zellen korreliert ist.
Photonen, die im Winkel von weniger als 20⁰ nach vorne gestreut werden, erzeugen das Forward Scatter Signal (FSC); dieser Parameter entspricht der Zellgröße. Orthogonal gestreute Photonen, die in einem Winkel von 90⁰ erfasst werden, erzeugen das Side Scatter Signal (SSC); dieser Parameter korrespondiert mit der Granularität der Zelle. Zusätzlich können Fluoreszenzlichtsignale mittels Durchflusszytometrie, ebenfalls in einem Winkel von 90°, erfasst und durch eine Reihe von Filtern geleitet werden, um spezifische Fluoreszenzemissionen zu messen. Dies liefert diverse zellgebundene Informationen, die zur Unterscheidung verschiedener Zelltypen in Populationen oder Gemeinschaften einer Probe genutzt werden können, was für unser Umweltlabor von immenser Bedeutung ist.
Die Zellsortierung (FACS) wird zur präzisen Isolation einzelner Zellen eingesetzt. Bei dieser Methode werden stromabwärts der Detektionszone Tröpfchen gebildet, die die Zielzellen enthalten. Diese Tröpfchen werden dann entweder positiv oder negativ geladen und durch Ablenkplatten voneinander getrennt. Jedes spezifische Merkmal, wie Fluoreszenzintensität oder Zellgröße, kann für eine Sortierentscheidung herangezogen werden. Vitale Zellen können auf Agarplatten, in Wells oder Röhrchen mit geeigneten Wachstumsmedien sortiert werden. Es ist auch möglich, Zellen für weiterführende Downstream-Analysen zu sortieren, z.B. für die Metagenom-Sequenzierung spezifischer Zellsubpopulationen. Die gewonnenen Daten sind entscheidend, um beispielsweise die Auswirkungen von [kreuzfahrtschiffe umweltverschmutzung](https://de.viettopreview.vn/kreuzfahrtschiffe-umweltverschmutzung/) auf aquatische Mikrobiome zu verstehen oder Beiträge zum [umwelt schützen](https://de.viettopreview.vn/umwelt-schutzen/) zu leisten.
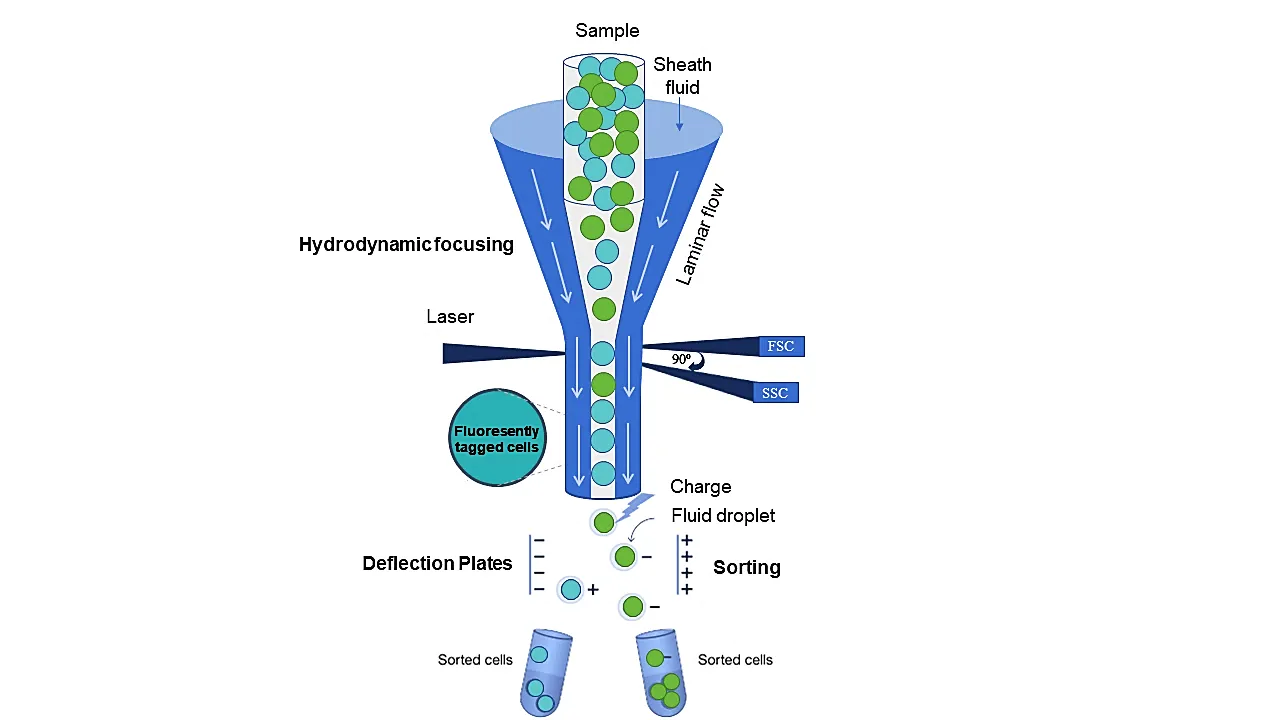 Principle of Flow Cytometry Das Prinzip der Durchflusszytometrie erklärt die Analyse von Zellen im Flüssigkeitsstrom.
Principle of Flow Cytometry Das Prinzip der Durchflusszytometrie erklärt die Analyse von Zellen im Flüssigkeitsstrom.
Modernste Ausstattung im Umweltlabor
Für verschiedene Forschungsaktivitäten stehen uns fünf Durchflusszytometer zur Verfügung. Drei davon werden für Sortierzwecke eingesetzt und zwei als Benchtop-Geräte. Eine vergleichende Übersicht unserer aktuellen Geräte finden Sie in Tabelle 1.
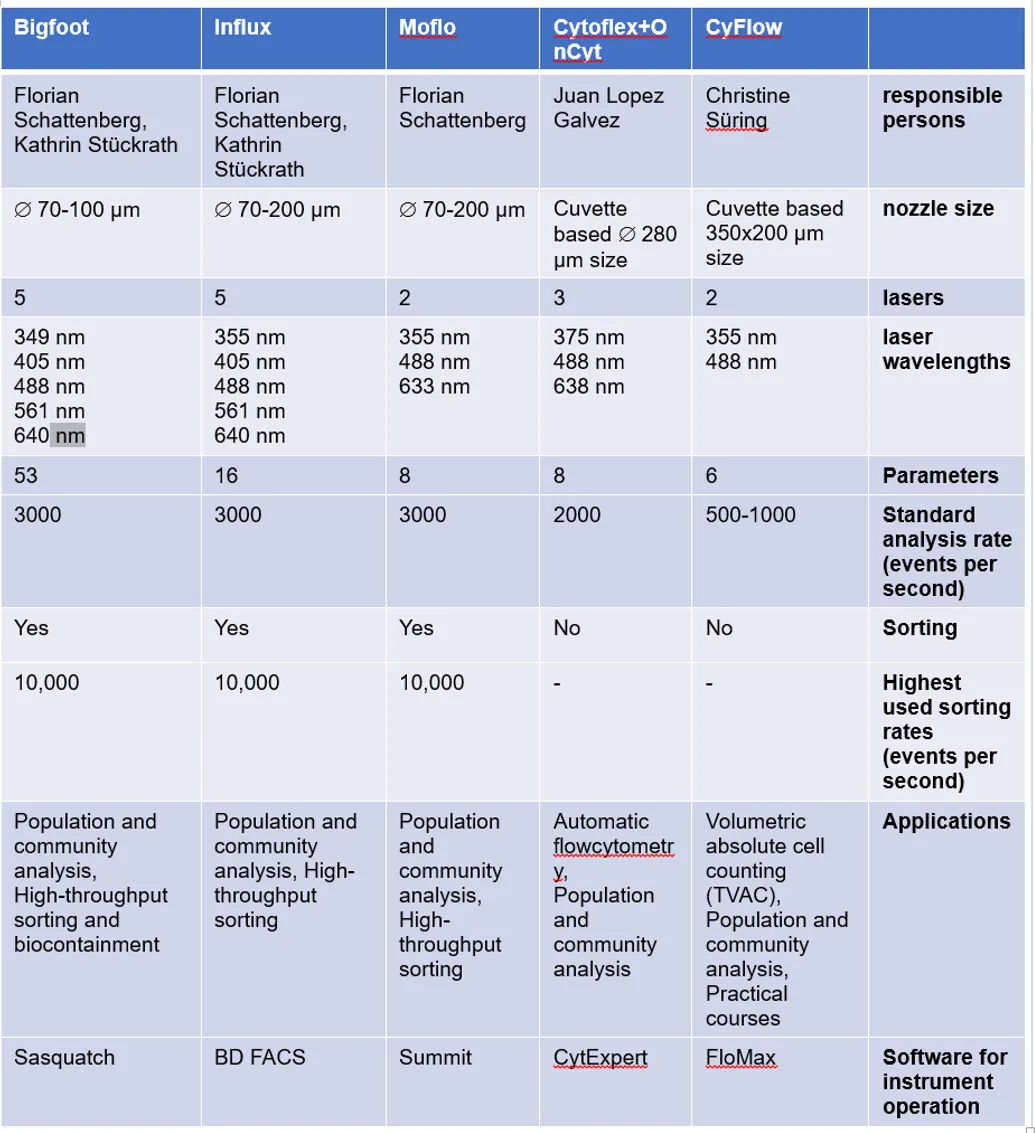 Equipment table Die Tabelle zeigt eine Übersicht der im Labor verfügbaren Durchflusszytometer.
Equipment table Die Tabelle zeigt eine Übersicht der im Labor verfügbaren Durchflusszytometer.
1. Bigfoot
Mit bis zu 5 Lasern und 53 Detektoren bietet das Bigfoot-Gerät spektrale Entmischungsfähigkeiten, die Zelltypen in Populationen und Gemeinschaften auf hohem Niveau unterscheiden können. Es erkennt Autofluoreszenz und entsprechende Veränderungen definierter und sensibler als andere Geräte. Die Biosicherheitsstufe 2 (BSL-2) mit integriertem HEPA-Filter und Aerosolmanagement sorgt für integrierte Biokontaminationseindämmung und Personenschutz. Dies macht Hochleistungs- und Hochdurchsatzmessungen für Downstream-OMICS-Anwendungen bequemer und schneller. Der Bigfoot Spectral Cell Sorter ermöglicht Multiway-Sortierung in 1,5 mL bis 50 mL Röhrchen, in 24- bis 384-Well-Plates und sogar in 1536-Well-Sortierplatten (Thermo Fisher Scientific. Bigfoot spectral cell sorter: https://www.thermofisher.com/de/de/home/life-science/cell-analysis/flow-cytometry/flow-cytometers/bigfoot-spectral-cell-sorter.html).
2. Influx
Der BD Influx™ Zellsorter verfügt über 5 Anregungslaser, die gleichzeitig mehrere Zellparameter erfassen können. Die Detektion geringer Fluoreszenzintensitäten ermöglicht es, Bakterien besonders gut zu charakterisieren und mikrobielle Populationen und Gemeinschaften auf hohem Niveau aufzulösen. Er verwendet die BD FACS™ Accudrop-Technologie, um eine präzise Tropfenverzögerung für die Sortierung direkt auf Kulturplatten, 24- bis 96-Well-Plates und 1,5–50 mL Röhrchen zu bestimmen. Mehrfache 4-Wege- und 6-Wege-Sortierungen sind möglich (https://www.bd.com/resource.aspx?IDX=17866).
3. MoFlo
Der MoFlo Legacy Zellsorter ist mit drei Lasern ausgestattet. Er wird sowohl für Subpopulations- als auch für Gemeinschaftsanalysen eingesetzt. Der MoFlo ist ein älteres Gerät, das demnächst abgebaut wird.
4. CyFlow
Im Vergleich zu anderen Zytometern ist der CyFlow Space relativ einfach zu bedienen und bietet eine Auflösung von 0,5 bis 100 µm für die Partikelmessung. Er analysiert die absolute Zell- und Partikelanzahl pro mL mittels True Volumetric Absolute Counting (TVAC). Er wird für Routineanalysen von Umweltproben wie Abwasser und für die Ausbildung von Studierenden in Praktika und Universitäts-Praktika eingesetzt (https://www.sysmex.de/produkte/details/cyflow-space.html). Dies unterstreicht die Relevanz des CyFlow als Kernstück in unserem Umweltlabor für die angewandte Forschung.
5. CytoFLEX
CytoFLEX (Beckman Coulter, Brea, CA, USA) enthält 3 Laser und ist mit einem OC-300 Automatisierungsgerät (onCyt Microbiology, Zürich, Schweiz) für automatisierte Durchflusszytometrie-Operationen wie Probenahme, Fixierung und Färbung gekoppelt. Das Gerät ist eine Leihgabe von OnCyte und wird nur bis Mitte 2025 verfügbar sein. Es misst die Zelldichte oder Fingerabdruck-ähnliche Muster bakterieller Populationen oder Gemeinschaften und generiert somit geeignete Daten für leistungsstarke automatische Datenanalyse- und Interpretationspipelines. Dies macht die Daten in kürzester Zeit verfügbar und ermöglicht die Online-Überwachung von Bioreaktoren.
 flow Cytometers Eine Übersicht der Durchflusszytometer im Labor.
flow Cytometers Eine Übersicht der Durchflusszytometer im Labor.
Methoden für umfassende Analysen im Umweltlabor
Proben
Die Durchflusszytometrie-Gruppe ist auf die Analyse von prokaryotischen (Bakterien, Cyanobakterien und Archaeen) und eukaryotischen (Hefe und Algen) Zellen spezialisiert. Mikroorganismen können in allen Lebensstadien analysiert werden, z.B. vegetative/tote Zellen, Sporen, Biofilme, etc. Suspensionen mit Einzelzellen sind dabei unerlässlich. Für alle Proben wird eine zusätzliche Filtration mit Celltrics-Filtern mit einer Porengröße von 50 µm durchgeführt, um ein Verstopfen der 70 µm Düse zu verhindern. Dies gewährleistet die Präzision und Zuverlässigkeit der Ergebnisse in unserem Umweltlabor.
Lebende versus fixierte Zellen
Die Durchflusszytometrie kann sowohl mit lebenden (Echtzeit-) als auch mit fixierten Zellen durchgeführt werden. Die Fixierung mit Chemikalien wie Paraformaldehyd (PFA) vernetzt intensiv Oberflächenproteine der Zellen, was deren Membranen stabilisiert und verschließt. Nach 30 Minuten wird das PFA entfernt und die Zellen in 70 % Ethanol suspendiert und bei -20°C gelagert. Diese Methode ermöglicht es uns, Proben über lange Zeiträume von bis zu 6 Monaten zu lagern. Darüber hinaus gibt es keine Biosicherheitsbedenken bei den fixierten Proben. Ähnliche Färbeprotokolle können sowohl für lebende als auch für fixierte Zellen verwendet werden, um spezifische zelluläre Komponenten oder Biomarker zu markieren. Lebende Zellen sind metabolisch aktiv und behalten ihre physiologischen Funktionen bei, während fixierte Zellen ihre Stoffwechselaktivität eingestellt haben und ihre Zellstruktur erhalten ist. Für die Fixierung von Zellen stehen zwei Fixierungsprotokolle zur Verfügung.
 Table2 Die Tabelle beschreibt verschiedene Fixierungsprotokolle für die Zellanalyse.
Table2 Die Tabelle beschreibt verschiedene Fixierungsprotokolle für die Zellanalyse.
Färbung
1. Fluoreszierende Proteine
Die fluoreszenzmarkierten Reporterproteine können einfach gemessen werden. Einige der weit verbreiteten fluoreszierenden Proteine umfassen:
A. Green Fluorescent Protein (GFP): GFP ist eines der am häufigsten verwendeten fluoreszierenden Proteine als Marker für die Genexpression in der Molekular- und Zellbiologie. Die Anregungs- und Emissionswellenlängen für GFP liegen bei 470 bis 490 nm bzw. 510 bis 530 nm.
B. Red Fluorescent Protein (RFP): RFPs, wie DsRed und mCherry, emittieren rotes Licht und werden oft zusammen mit GFP für Dual-Labeling-Experimente verwendet. RFPs haben typischerweise eine Anregungswellenlänge im Bereich von 550 bis 590 nm und eine Emissionswellenlänge um 590 bis 650 nm.
C. Yellow Fluorescent Protein (YFP): YFP, wie Citrin (abgeleitet von Aequorea victoria), emittiert gelbes Licht und wird häufig in Kombination mit anderen fluoreszierenden Proteinen verwendet. YFP hat eine Anregungswellenlänge typischerweise im Bereich von 505 bis 515 nm und eine Emissionswellenlänge typischerweise im Bereich von 520 bis 570 nm.
D. Cyan Fluorescent Protein (CFP): CFPs, wie Cerulean (abgeleitet von Aequorea victoria), emittieren cyanfarbenes Licht und werden in mehrfarbigen Bildgebungs-Experimenten eingesetzt. CFP hat eine Anregungswellenlänge von etwa 433 nm und eine Emissionswellenlänge von etwa 475 nm.
E. Blue Fluorescent Protein (BFP): BFPs emittieren blaues Licht und können in Kombination mit anderen fluoreszierenden Proteinen für mehrfarbige Bildgebung verwendet werden. BFPs haben eine Anregungswellenlänge im Bereich von etwa 380 bis 410 nm und eine Emissionswellenlänge um 440 bis 470 nm.
2. Autofluoreszenz
Einige Bakterien, Cyanobakterien, Algen und Archaeen enthalten endogene Fluorophore, die bei Anregung durch UV-/Sichtbare Strahlung einer geeigneten Wellenlänge Autofluoreszenz emittieren. Verschiedene zelluläre Komponenten können zur Autofluoreszenz beitragen, einschließlich Pigmente. Die auf Autofluoreszenz basierende Identifizierung ermöglicht eine markierungsfreie Detektion, ohne dass exogene fluoreszierende Marker oder Sonden erforderlich sind.
3. Fluoreszierende Farbstoffe
Zellen können durch eine Vielzahl von fluoreszierenden Reagenzien für die Durchflusszytometrie-Analyse angefärbt werden. Dazu gehören DNA-bindende Farbstoffe, Vitalitätsfarbstoffe und Ionenindikatorfarbstoffe. In der Durchflusszytometrie-Gruppe werden die folgenden fluoreszierenden Farbstoffe für die Populations- und Gemeinschaftsanalyse verwendet:
- DAPI (4′,6-Diamidino-2-phenylindol): DAPI ist ein DNA-spezifischer Fluoreszenzfarbstoff, der zur Anfärbung bakterieller DNA verwendet werden kann. Er wird häufig zur Beurteilung des Zellzyklusfortschritts und des Community-Fingerprintings eingesetzt.
- SYTO 9: SYTO-Farbstoffe sind Nukleinsäurefärbemittel, die intakte Bakterienzellen durchdringen können. Sie werden oft für die Gesamtzellzählung und Vitalitätsbeurteilungen verwendet. SYTO 9 ist ein grün-fluoreszierender Farbstoff, der sowohl lebende als auch tote Zellen anfärbt.
- Propidiumiodid (PI): PI ist ein DNA-bindender Farbstoff, der zur Unterscheidung zwischen lebenden und möglicherweise toten Zellen basierend auf ihrer Membranintegrität verwendet werden kann. Lebende Zellen schließen PI aus, während Zellen mit beeinträchtigten Membranen PI eindringen lassen.
- SYBR Green: SYBR Green ist ein Nukleinsäure-bindender Farbstoff, der Fluoreszenz bei Bindung an doppelsträngige Nukleinsäuren emittiert. SYBR Green bindet sowohl an DNA als auch an RNA.
- Hoechst 33342: Hoechst 33342 ist ein zellpermeabler DNA-Farbstoff, der bevorzugt an Adenin-Thymin (AT)-reiche Regionen der DNA bindet. Er wird zur Quantifizierung des DNA-Gehalts und zur Identifizierung verschiedener Phasen des Zellzyklus verwendet.
- FDA (Fluoresceindiacetat) ist ein nicht-fluoreszierender, zellpermeabler Farbstoff, der in lebenden Zellen über intrazelluläre Esterasen in ein grün fluoreszierendes Produkt, Fluorescein, umgewandelt wird. Tote Zellen zeigen keine Esteraseaktivität, daher wird der Farbstoff zur Beurteilung der Zellvitalität verwendet.
- 3,3′-Dihexyloxacarbocyaniniodid (diOC6(3)) ist ein lipophiler kationischer Farbstoff, der Veränderungen im bakteriellen Membranpotenzial anzeigt und Einblicke in die bakterielle Vitalität, Membranpotenzialaktivität und Reaktionen auf verschiedene Umweltbedingungen geben kann.
- Nilrot ist ein lipophiler Farbstoff, der weit verbreitet zur Anfärbung und Visualisierung neutraler Lipide, insbesondere Triglyceride und Lipoproteine, in Zellen verwendet wird.
- Lektine oder Antikörper, die mit fluoreszierenden Farbstoffen konjugiert sind, können zur Identifizierung spezifischer Zellpopulationen basierend auf kohlenhydratvermittelten Interaktionen oder zur Bindung an spezifische Zelloberflächenregionen verwendet werden.
Die präzise Anwendung dieser Färbemethoden ist entscheidend für die hochwertigen Analysen, die unser Umweltlabor bietet, und trägt dazu bei, selbst komplexe Fragestellungen wie die mikrobielle Reaktion auf [lng umwelt](https://de.viettopreview.vn/lng-umwelt/) zu beleuchten.
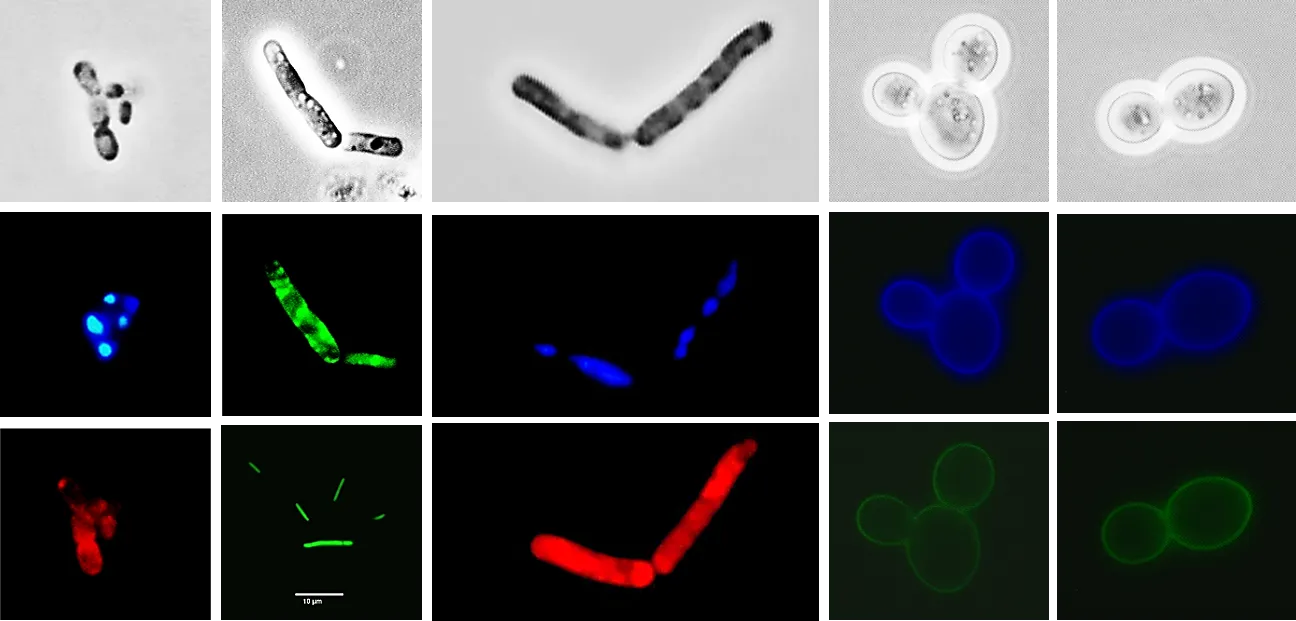 Microscopy Mikroskopische Aufnahmen von angefärbten Zellen.
Microscopy Mikroskopische Aufnahmen von angefärbten Zellen.
Ausrichtung der Durchflusszytometer
In der Durchflusszytometrie können Ungenauigkeiten durch unpräzise Ausrichtung der Optik und Schwankungen der Laserstabilität über die Zeit entstehen. Um dies zu vermeiden, werden Laser sorgfältig ausgerichtet, um sicherzustellen, dass ihre Strahlen korrekt auf den Probenstrom fokussiert sind, um die Fluoreszenzemission zu detektieren. Standardisierte monodisperse Beads werden routinemäßig in den Durchflusszytometern eingesetzt. Typischerweise werden mono- oder multifluoreszierende Beads mit einer Größe von 0,5 bis 1 μm für diesen Zweck verwendet. Diese Kalibrierungsbeads sind fluoreszierende Mikrosphären mit bekannten Fluoreszenzeigenschaften, einschließlich Fluoreszenzintensität und Emissionsspektren. Durch die Analyse von Kalibrierungsbeads im Durchflusszytometer können Operatoren die PMT-Spannungen und Verstärkungseinstellungen anpassen, um die Signal-Rausch-Verhältnisse zu standardisieren und zu optimieren. Eine ordnungsgemäße Ausrichtung mittels Kalibrierungsbeads verbessert die Zuverlässigkeit und Reproduzierbarkeit von Durchflusszytometriedaten und ermöglicht eine genaue Quantifizierung und Analyse von Proben, die für unser Umweltlabor von größter Bedeutung ist.
Künstliche Mikrobengemeinschaft (mCMC)
Während Ausrichtungsbeads dazu beitragen, instrumentelles Rauschen zu minimieren und die allgemeinen Maschineneinstellungen zu harmonisieren, ist die Verwendung einer künstlichen mikrobiellen zytometrischen Mock-Gemeinschaft (mCMC) unverzichtbar zur Validierung von Labor-Workflows, Färbeprotokollen und zum Datenvergleich über Experimente hinweg. Die mCMC dient als wichtige Referenzstandard für die konsistente Charakterisierung mikrobieller Gemeinschaften mittels zytometrischer Analyse, insbesondere in Langzeitstudien.
Die mCMC, bestehend aus drei reinen Stämmen (Stenotrophomonas rhizophila DSM 14405, Kocuria rhizophila DSM 348 und Paenibacillus polymyxa DSM 36), weist Zellpopulationen auf, die im 2D-Plot über den unteren und oberen logarithmischen Skalenbereich verteilt sind, was ihre Trennung durch Gating ermöglicht. Darüber hinaus adressiert die mCMC die Heterogenität, die durch Subpopulationen entsteht, und fügt der Analyse Komplexität hinzu. Ihr Nutzen erstreckt sich auf die Anpassung der Bakterienposition und des Auflösungsbereichs in allen kommerziellen Durchflusszytometern, wodurch die Instrumentenauflösung verbessert und Hintergrundrauschen reduziert wird. Sie hilft auch, bedienerabhängige Fehler oder Variationen in der Zellbehandlung zu identifizieren und zu mildern, und konsistente Zellpositionen während Langzeitexperimenten aufrechtzuerhalten, um die Stabilität zytometrischer Gemeinschaftsmuster zu gewährleisten und somit automatisierte bioinformatische Auswertungs-Pipelines zu erleichtern.
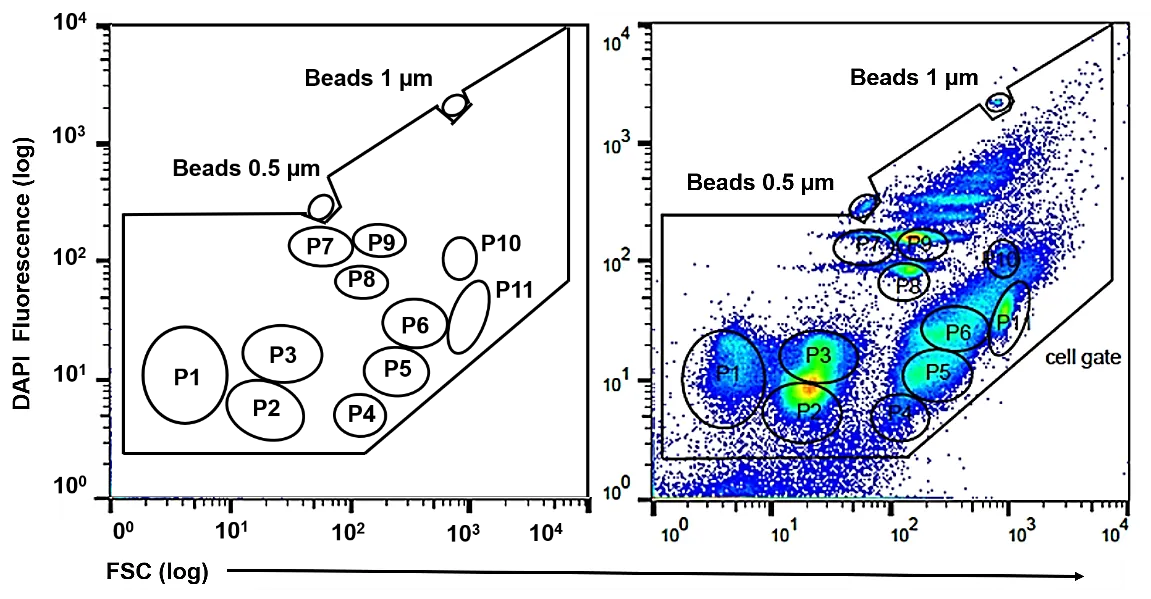 Mock community Zellgate und Gatemuster der Platten-mCMC. Der 2D-Plot (rechts) zeigt die konstruierte Mock-Gemeinschaft (P. polymyxa (P10 und P11), S. rhizophila (P1 bis P3) und K. rhizophila (P4 bis P6) im Verhältnis von 80:1:19). (https://www.nature.com/articles/s41596-020-0362-0)
Mock community Zellgate und Gatemuster der Platten-mCMC. Der 2D-Plot (rechts) zeigt die konstruierte Mock-Gemeinschaft (P. polymyxa (P10 und P11), S. rhizophila (P1 bis P3) und K. rhizophila (P4 bis P6) im Verhältnis von 80:1:19). (https://www.nature.com/articles/s41596-020-0362-0)
Bioinformatische Analysetools im Umweltlabor
Um künstliche oder natürliche Gemeinschaften in gemanagten Systemen zu untersuchen, zu überwachen und schließlich zu kontrollieren, sind schnelle Detektions- und Bewertungsmethoden erforderlich. Zytometrische Fingerabdrücke komplexer mikrobieller Gemeinschaften und Reinkultur-Subpopulationen können über die grafische Benutzeroberfläche (GUI) des biTCa Analyze Tools ausgewertet werden. Es enthält alle von unserem Labor entwickelten bioinformatischen Tools wie Cytometric Histogram Image Comparison (flowCHIC), Cytometric Barcoding (flowCyBar) und flowEMMi. Diese Tools sind integraler Bestandteil unseres Umweltlabor-Ansatzes, um die enorme Datenmenge effizient zu verarbeiten.
1. Cytometric Histogram Image Comparison (flowCHIC)
Alle zytometrischen 2D-Plots werden in Graustufenbilder umgewandelt, und diese Bilder werden dann basierend auf dem durchschnittlichen Grauwert pro informativem Pixel verglichen. Die Unterschiede in der Zellhäufigkeit werden durch die NMDS-Plots dargestellt. Das R-Paket flowCHIC und seine Bedienungsanleitung können von der Bioconductor-Plattform heruntergeladen werden als Schumann J, Koch C, Fetzer I, Müller S (2019). flowCHIC – Analyze flow cytometric data of complex microbial communities based on histogram images. R package version 1.36.0, https://bioconductor.org/packages/flowCHIC.
https://www.frontiersin.org/journals/microbiology/articles/10.3389/fmicb.2014.00273/full
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6126456/
https://onlinelibrary.wiley.com/doi/full/10.1002/cyto.a.22286
2. Cytometric Barcoding (CyBar)
CyBar illustriert die Veränderungen in der Zellhäufigkeit von Subgemeinschaften in Korrelation zu zeitlichen, räumlichen, abiotischen und biotischen Parametern mittels NMDS, Boxplots und Heatmaps (Korrelation, Interaktionspotenzial). Das R-Paket flowCyBar und seine Bedienungsanleitung können von der Bioconductor-Plattform heruntergeladen werden als Schumann J, Koch C, Günther S, Fetzer I, Müller S (2015). flowCyBar – Analyze flow cytometric data using gate information. R package version 1.38.0, https://bioconductor.org/packages/flowCyBar.
https://pubs.acs.org/doi/full/10.1021/es3041048
https://www.pnas.org/doi/10.1073/pnas.2117814119
https://www.nature.com/articles/nprot.2012.149
3. flowEMMi
flowEMMi v2 ist ein automatisches modellbasiertes Clustering-Tool für zytometrische Gemeinschaftsdaten, das auf multivariaten Gaußschen Mischmodellen basiert. Es markiert alle Subgemeinschaften in den Histogrammen mit verschiedenen Gates, und eine kombinierte Gate-Vorlage wird schließlich auf alle Proben angewendet. Dieses Tool bietet schnelles und genaues Zell-Clustering für dynamische Gemeinschaftsanalysen, wobei technisches Rauschen oder Beads umgangen werden.
flowEMMi v2: Software kann heruntergeladen und installiert werden von https://github.com/choener/flowEMMi
https://www.sciencedirect.com/science/article/pii/S200103702200527X
https://www.mdpi.com/2073-4409/12/12/1559
Anwendungen unserer Forschung im Umweltlabor
1. Mikrobielle Gemeinschaftsökologie
Die Aufrechterhaltung der gewünschten mikrobiellen Gemeinschaft in Bioreaktoren und die Bestimmung der Strukturen von Gemeinschaften in natürlichen Biosphären sind wichtige Anwendungen der mikrobiellen Durchflusszytometrie. Mikrobielle Gemeinschaften können hochdivers sein, sind anfällig für stochastische Fluktuationen, und der Verlust von Schlüsselakteuren ist häufig. Unsere durchflusszytometrische Analyse in Kombination mit bioinformatischen und ökologischen Tools offenbart die Dynamik und das Schicksal von Gemeinschaftsstrukturen viel schneller als Sequenzierungstechnologien. Wir verwenden verschiedene Diversitätsindizes und Interaktionsprofile, um Gemeinschafts-Assemblierungsstrategien und die funktionellen Abhängigkeiten von Gemeinschaftsmitgliedern zu beschreiben. Dieses Fachwissen bieten wir an, um das ökologische Paradigma zu verstehen, das die interessierenden Gemeinschaften in ihren jeweiligen Umgebungen bildet. So können wir beispielsweise die Umweltauswirkungen der [umweltverschmutzung kreuzfahrtschiffe](https://de.viettopreview.vn/umweltverschmutzung-kreuzfahrtschiffe/) auf marine Mikrobiome genauer untersuchen.
Zu beantwortende Fragen:
- Analyse der internen Strukturvariation mikrobieller Gemeinschaften und Identifizierung von Subgemeinschaftsfunktionen.
- Ermöglichung der Quantifizierung von Einzelzelldynamiken und der Online-Bewertung von Gemeinschaftsattributen.
- Berechnung der Stabilitätseigenschaften Resistenz, Resilienz, Verdrängungsgeschwindigkeit und Elastizität der Bioreaktor-Gemeinschaft.
- Interpretation der Gemeinschaftsparadigmen, Trends und Entwicklungen.
- Aufdeckung möglicher Stimuli und Einschränkungen, die die Gemeinschaftsstruktur und -funktion beeinflussen.
- Aufrechterhaltung stabiler Gemeinschaften in Ihrem Bioreaktor, Vermeidung von Auswaschung.
Vorgehensweise:
Zellen müssen fixiert werden. Die Zelldichte muss angepasst, DAPI-Färbung durchgeführt und ein Fingerprinting vorgenommen werden. Die resultierenden Subgemeinschaften müssen gegatet und die Zellfrequenz pro Gate bestimmt werden. Jedes Bewertungstool kann dann verwendet werden.
Anforderungen an die Probe:
Die Probe muss eine Zellsuspension ohne Verunreinigungen sein. Die Zellen können sonifiziert, gevortexed, gewaschen und filtriert werden, um Aggregate und Partikel zu entfernen.
Entwickelte Technologien:
A. Looped Mass Transfer Technologie: Unsere kürzlich entwickelte Looped Mass Transfer Technologie stellt sicher, dass die mikrobiellen Gemeinschaften in den Bioreaktoren stabil gehalten und zurückgehalten werden. Wenn Sie mit dem Verlust mikrobieller Gemeinschaften in Ihrem Bioprozess zu kämpfen haben, könnte dies eine alternative Lösung sein. Kontaktieren Sie uns.
B. Stabilitäts- und Diversitätsmessungen: Ökologische Stabilitätseigenschaften mikrobieller Gemeinschaften können mittels Durchflusszytometrie bewertet werden.
C. Nettowachstumsrate: Die Bestimmung des Wachstums von Subpopulationen innerhalb von Gemeinschaften kann mittels Durchflusszytometrie bewertet werden.
https://www.pnas.org/doi/full/10.1073/pnas.2117814119
https://enviromicro-journals.onlinelibrary.wiley.com/doi/10.1111/1462-2920.13402
https://experiments.springernature.com/articles/10.1038/nprot.2012.149
https://pubmed.ncbi.nlm.nih.gov/30289191/
https://onlinelibrary.wiley.com/doi/10.1002/cyto.a.23965
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5770544/
https://onlinelibrary.wiley.com/doi/10.1002/eji.201646297
https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-020-0788-1
https://microbialcellfactories.biomedcentral.com/articles/10.1186/s12934-019-1143-8
2. Differentielle Färbung und Subpopulationsanalyse
Die Biokonversion könnte durch die Analyse verschiedener Subpopulationen und ihrer Beiträge verbessert werden. Dies ermöglicht die Prozessoptimierung durch Änderung operativer Parameter. Für die differenzierte Färbung von Zellen aus Rein- oder Kokulturen steht eine Reihe fluoreszierender Farbstoffe zur Verfügung für:
- Biomolekülsynthese
- Metabolisch aktive Zustände
- Zellzyklusstudien
- Subpopulationsanalyse
Zu beantwortende Fragen:
- Untersuchung verschiedener physiologischer oder expressioneller Subpopulationen des Organismus von Interesse.
- Zellzyklusmerkmale des Organismus in Abhängigkeit von verschiedenen Substraten oder Einschränkungen.
- Vitalität und Zählung lebender/toter Zellen.
- Polymerbildung wie Polyhydroxyalkanoate (PHA).
- Fluoreszenzmarkierte Genexpression in Subpopulationen.
Vorgehensweise:
Zellen können lebend oder fixiert gemessen werden (mit den etablierten Fixierungsprotokollen). Nach optischer Dichteanpassung, Färbung und Filtration werden die Zellen zytometrisch analysiert.
Anforderungen an die Probe:
Die Probe muss eine Zellsuspension ohne Verunreinigungen sein, die sonifiziert, gevortexed, gewaschen und filtriert werden kann, um Aggregate und Partikel zu entfernen. Die Forschung in unserem Umweltlabor ist hier entscheidend, um beispielsweise die mikrobiellen Prozesse bei der [zement herstellung umwelt](https://de.viettopreview.vn/zement-herstellung-umwelt/) zu analysieren und gegebenenfalls nachhaltigere Alternativen zu entwickeln.
https://onlinelibrary.wiley.com/doi/epdf/10.1111/j.1365-2184.2007.00465.x
https://onlinelibrary.wiley.com/doi/pdf/10.1002/cyto.a.20402
https://enviromicro-journals.onlinelibrary.wiley.com/doi/10.1111/1751-7915.12018
https://www.sciencedirect.com/science/article/pii/S0958166912001723
https://pubs.acs.org/doi/10.1021/ac501118v
3. Sortierung für OMICS-Anwendungen
Die Arbeitsgruppe Durchflusszytometrie verfügt über drei Sortierer. Einzelzellen können aufgrund ihrer Fluoreszenzeigenschaften voneinander unterschieden und sortiert werden. Ausgewählte Zellen werden für eine Reihe von nachgeschalteten Experimenten verwendet, einschließlich funktioneller Assays, DNA-Analyse, Gen-/Proteinexpressionsanalyse, Reinkulturen, Koloniecharakterisierung und Stammentwicklung.
Zu beantwortende Fragen:
- Trennung von fluoreszenzemittierenden Reporterzellen, die das Protein von Interesse exprimieren, von Hintergrundzellen.
- Sortierung von differenziell gefärbten Zellen voneinander und von nicht gefärbten Zellen.
- Sortierung verschiedener physiologischer Zustände von Zellen während der Replikation zur Rekultivierung.
- Trennung und Sortierung von gering abundanten Zellen von hoch abundanten Zellen für 16S rRNA-Sequenzierung oder Gesamtgenom-Sequenzierung oder andere OMICS-Anwendungen.
- Sortierung verschiedener Zellen und Sporen basierend auf Größe und Dichte.
- Sortierung von Archaeen/Cyanobakterien von Bakterien basierend auf ihrer Autofluoreszenz.
- Sortierung lebender Zellen von toten Zellen.
Vorgehensweise:
Identifizierung des Sortierbereichs durch Gating. Bitte kontaktieren Sie die verantwortliche Person, um erste Testläufe zu vereinbaren.
Anforderungen an die Probe:
Die Probe muss eine Zellsuspension mit Zelldichten von nicht weniger als 10⁵ sein. Die Zellen können sonifiziert, gevortexed, gewaschen und filtriert werden, um Aggregate und Verunreinigungen zu entfernen. Das Volumen hängt von den nachgeschalteten Anwendungen ab.
https://www.mdpi.com/2076-2607/11/1/175
https://microbialcellfactories.biomedcentral.com/articles/10.1186/s12934-016-0610-8
https://microbialcellfactories.biomedcentral.com/articles/10.1186/s12934-017-0793-7
Fazit: Das Umweltlabor als Vorreiter der mikrobiellen Forschung
Das Umweltlabor der Durchflusszytometrie-Gruppe bietet eine unverzichtbare Infrastruktur und Expertise für die moderne mikrobielle Forschung. Durch die Kombination modernster Durchflusszytometer mit präzisen Färbemethoden und fortschrittlichen bioinformatischen Tools sind wir in der Lage, komplexe mikrobielle Gemeinschaften und Zellpopulationen auf einer bisher unerreichten Ebene zu analysieren und zu verstehen.
Unsere Fähigkeiten erstrecken sich von der detaillierten Charakterisierung einzelner Zellen bis hin zur Aufklärung der Dynamik ganzer Ökosysteme, wie sie in Bioreaktoren oder natürlichen Umgebungen vorkommen. Dies ermöglicht nicht nur grundlegende wissenschaftliche Erkenntnisse, sondern auch die Entwicklung praktischer Lösungen, sei es zur Optimierung von Bioprozessen, zur Überwachung der Umweltqualität oder zur Identifizierung von Schlüsselakteuren in der biologischen Vielfalt.
Als führendes Umweltlabor sind wir stets bestrebt, die Grenzen des Möglichen in der Durchflusszytometrie zu erweitern und wertvolle Beiträge zu einem tieferen Verständnis mikrobieller Lebenswelten zu leisten. Wenn Sie an einer Zusammenarbeit interessiert sind oder spezifische Analyseanforderungen haben, zögern Sie nicht, uns zu kontaktieren.
